ggcor【安装方案实测成功】-程序员宅基地
致敬大佬:
画图网站:ImageGP | ImageGP (bic.ac.cn)
BIC - Bioinfo Intelligent Cloud
这里要介绍的ggcor是corrplot的有一种实现,在吸收借鉴(或者说是全般)corrplot的基础上,略有提升,使用上会更灵活简单。
相关系数矩阵可视化已经至少有两个版本的实现了,魏太云基于base绘图系统写了corrplot包,应该说是相关这个小领域中最精美的包了,使用简单,样式丰富,只能用惊艳来形容。Kassambara的ggcorrplot基于ggplot2重写了corrplot,实现了corrplot中绝大多数的功能,但仅支持“square”和“circle”的绘图标记,样式有些单调,不过整个ggcorrplot包的代码大概300行,想学习用ggplot2来自定义绘图函数,看这个包的源代码很不错。还有部分功能相似的corrr包(在写ggcor之前完全没有看过这个包,写完之后发现在相关系数矩阵变data.frame方面惊人的相似),这个包主要在数据相关系数提取、转换上做了很多的工作,在可视化上稍显不足。ggcor的核心是为相关性分析、数据提取、转换、可视化提供一整套解决方案,目前的功能大概完成了70%,后续会根据实际需要继续扩展。
方法:
1、在git hub上找到源(https://github.com/mj163163/ggcor-1),并下载zip压缩包其他方案:
2、将zip压缩包放到到R的library文件夹内(这个要看自己保存在哪里了,我的是C:/Users/syg/Documents/R/win-library/4.0,其中4.0是R的版本号)。然后将压缩包名改为R可以识别的格式(我改成了ggcor_master)。
3、打开Rstudio,运行以下代码。注意:安装devtools时,如果弹出对话框选择“yes”;安装ggcor时,按enter跳过更新。
#安装devtools
install.packages("devtools")
#安装ggcor
devtools::install_local("C:/Users/syg/Documents/R/win-library/4.0/ggcor_master.zip")
devtools::install_github("zlabx/ggcor")
ggcor
一种检验两个矩阵相关关系的方法——Mantel test方法,这种分析方法可用在植物微生物群落与生态环境之间相关性的检验以及人体肠道微生物群落与人体疾病相关性检验等领域。
The goal of ggcor is to to provide a set of functions that be used to visualize simply and directly a correlation matrix based on ‘ggplot2’.
Installation
Now ggcor is not on cran, You can install the development version of ggcor from GitHub with:
# install.packages("devtools")
devtools::install_github("houyunhuang/ggcor")
数据预处理函数
ggplot2要求的数据格式是data.frame,要把相关系数矩阵处理成理想的数据格式需要一系列的操作。ggcor提供了两个主要的函数,一个是as_cor_tbl()函数,另一个是fortify_cor()函数,两者结合应该能满足绝大多数的应用场景需求。
as_cor_tbl()是更底层的函数,fortify_cor()本质上调用了as_cor_tbl()来得到最终的数据结果。这个函数适用于已经知道,或者需要用其它更特殊的函数(非stats::cor())来处理得到系数的情况,常用的参数是前三个。
-
x—— 相关系数矩阵(或者数据框),矩阵行名和列名是必要的,若没有或者缺失值会自动补全名字,行名以“Y”开头,附上递增的整数序列,列名以“X”开头,附上附上递增的整数序列。 -
type—— 相关系数矩阵图样式,“upper”截断下三角,“lower”截断上三角。 -
show.diag—— 相关系数矩阵图中是否包含对角线,仅对对称矩阵有效。 -
p—— 相关系数检验p值矩阵(或者数据框),必须与x一一对应。 -
low—— 相关系数置信区间下界矩阵(或者数据框),必须与x一一对应。 -
upp—— 相关系数置信区间上界矩阵(或者数据框),必须与x一一对应。 -
cluster.type—— 是否对相关系数矩阵进行重新排序,“none”表示不重排,“all”表示行列均重排,“row”表示对行重排,“col”则只对列重排。 -
...—— 其它传递给matrix_order()函数的参数。
library(ggcor)
## function(x,
## type = c("full", "upper", "lower"),
## show.diag = TRUE,
## p = NULL,
## low = NULL,
## upp = NULL,
## cluster.type = c("none", "all", "row", "col"),
## ...)
corr <- cor(mtcars)
df <- as_cor_tbl(corr)
df ## return a tibble
fortify_cor()主要适用于处理原始数据表,即调用cor()求相关系数,cor函数对数据按列进行两两相关性计算,默认使用pearson方法,当然理论解读中提前的spearman和kendall方法也都支持。调用(若需要)cor.test()函数进行统计检验,并返回ggcor需要的数据类。
-
x—— 原数据矩阵(或者数据框),列名是必要的,若没有或者缺失值会自动补全名字,列名以“X”开头,附上附上递增的整数序列。 -
y—— 原数据矩阵(或者数据框),列名是必要的,若没有或者缺失值会自动补全名字,列名以“X”开头,附上附上递增的整数序列。当y不为空(NULL)时,相关系数是x中的每一列和y中的每一列的相关性。 -
type—— 相关系数矩阵图样式,“upper”截断下三角,“lower”截断上三角。 -
show.diag—— 相关系数矩阵图中是否包含对角线,仅对对称矩阵有效。 -
cor.test—— 逻辑值,是否进行相关性检验。 -
cor.test.alt—— 相关性检验备择假设,详细请查看cor.test()帮助。 -
cor.test.method—— 相关性检验方法,详细请查看cor.test()帮助。 -
cluster.type—— 是否对相关系数矩阵进行重新排序,“none”表示不重排,“all”表示行列均重排,“row”表示对行重排,“col”则只对列重排。 -
cluster.method—— 当cluster.order为“HC”(默认)时算法,详细请查看ggcor::matrix_order()。 -
...—— 其它传递给cor()函数的参数。
## function(
## x,
## y = NULL,
## type = c("full", "upper", "lower"),
## show.diag = FALSE,
## cor.test = FALSE,
## cor.test.alt = "two.sided",
## cor.test.method = "pearson",
## cluster.type = c("none", "all", "row", "col"),
## cluster.method = "HC",
## ... )
df01 <- fortify_cor(mtcars, cor.test = TRUE, cluster.type = "all")
df01
as_cor_tbl()和fortify_cor()返回值均是cor_tbl类的数据框(准确的说是tibble),并包含其它额外特殊属性,要充分利用ggcor包中一系列工具函数带来的便捷性,必须调用(手动比较麻烦)这两个函数预处理数据。
初始化绘图函数
ggcor封装了一个基本的初始化函数ggcor(),用来处理数据、绘图类型、背景、坐标轴、颜色映射、图例等。看上去ggcor()函数非常复杂,但若仔细观察参数命名规则和分类,又是非常简单的。绘图区网格线参数名字均以grid.*开头;坐标轴相关参数均以axis.*开头;图例相关(主要是colourbar)的参数均以legend.*开头;还有panel.backgroud、xlim、ylim均是常见的参数,panel.backgroud参数用来设置绘图区背景颜色,xlim、ylim则是设置x/y轴的范围。把这几大块参数去掉后,剩下的参数只有6个了,瞬间清爽了不少。
要完全理解ggcor()的作用原理及相关参数的设置,需要先讲讲ggcor()内部构成。ggcor()本质上是调用了ggplot()来初始化,然后根据相关系数图的样式添加了一些辅助的图层。
-
x、y、mapping、is.cor、show.diag和...参数均和数据预处理和映射相关。 -
-
x可以是cor_tbl、矩阵(数据框)。当为cor_tbl时直接作为data参数传递给ggplot();为矩阵(数据框)时,若是(is.cor = TRUE)相关系数矩阵(数据框)时,调用as_cor_tbl()函数处理成cor_tbl,若不是(is.cor = FALSE)相关系数矩阵(数据框)时,调用fortify_cor()函数处理成cor_tbl,此时x、y、show.diag和...均传递给as_cor_tbl()或者fortify_cor()。 -
mapping对应ggplot()中的mapping参数,当为空(默认)时,根据cor_tbl中的变量情况添加,基础形式是aes(x = x, y = y, r = r, fill = r)。若cor_tbl包含“p”(进行了相关系数显著性检验),则最基础形式基础上额外添加p = p,若检验方法(cor.test.method = "pearson"),再加上low = low和upp = upp。
-
-
fill.*均是fill颜色映射函数相关的参数。 -
-
若
fill.scale.add为FALSE不添加颜色映射函数。若为TRUE(默认),则会在初始化中自动添加颜色映射函数。 -
fill.colours是颜色字符向量,用来控制填充色(fill)的颜色映射,若为空(默认),使用corrplot默认配色,可以通过ggcor:::.defualt.colors查看具体颜色。 -
fill.bin是否对连续性颜色进行分组(默认FALSE)。当fill.scale.add = TRUE时,若fill.bin = TRUE,ggcor()的颜色映射函数是使用scale_fill_steps2n(),若fill.bin = FALSE,ggcor()的颜色映射函数是使用scale_fill_gradient2n()。
-
-
背景网格线是通过
geom_tile()(目前来看,geom_tile()会存在一些难以处理的缺点,以后可能会添加专用的网格线图层函数),panel.backgroud(fill)、grid.*系列参数传递给geom_tile()。 -
legend.position传递给theme()中对应的参数,用来控制图例位置,其它legend.*开头参数传递给guide_colourbar()或者guide_colorsteps()。legend.breaks用来控制图例颜色棒标签显示位置,legend.labels是对应的标签。若fill.bin = TRUE,legend.breaks也是图例颜色棒切割分组的位置。 -
coord.fixed逻辑值,为真xlim、ylim传递给coord.fixed()函数,为假传递给coord_cartesian()函数。在ggcor包中,相关系数矩阵若是n * m的矩阵,那么第i行对应的坐标点(即as_cor_tbl()返回结果中的y)为n-i(为了和表格呈现样式一致,行方向翻转了),第j列对应的坐标点(即as_cor_tbl()返回结果中的x)为j,得到第(i, j)个数据点所在的方格坐标为(xmin = j-0.5, xmax = j+0.5, ymin = n-i-0.5, ymax = n-i+0.5)。从这个规律我们不难算出默认的xlim、ylim取值范围。注意,对cor_tbl数据框,或者说是ggcor包中函数操作数据,均不会改变每个数据单元格在图中的坐标位置。
ggcor()初始化之后,本质上返回的是ggplot对象,若是想改变默认设置,可以按照ggplot2的相应的函数和设置方法去调整。
## function(
## x,
## y = NULL,
## mapping = NULL,
## is.cor = FALSE,
## show.diag = TRUE,
## fill.scale.add = TRUE,
## fill.colours = NULL,
## fill.bin = FALSE,
## panel.backgroud = NA,
## grid.colour = "grey50",
## grid.size = 0.25,
## grid.linetype = "solid",
## axis.x.position = c("auto", "bottom", "top"),
## axis.y.position = c("auto", "left", "right"),
## fill.colours = NULL,
## legend.title = "correalation",
## legend.position = "auto",
## legend.breaks = NULL,
## legend.labels = NULL,
## coor.fixed = TRUE,
## xlim = NULL,
## ylim = NULL,
## ...)ggcor(mtcars)
ggcor(mtcars, type = "lower")
df02 <- fortify_cor(mtcars, type = "upper")
ggcor(df02, panel.backgroud = "#66C2A5")图层函数
ggcor提供了定制的geom_square()、geom_circle2()、geom_ellipse2()、geom_pie2()、geom_colour()、geom_confbox()、geom_num()、geom_mark()、geom_cross()9个ggplot2图层函数,可以根据需要进行叠加。除了ggplot2中一般化的参数(x、y、fill、colour、size等)最常用参数r、p、low、upp、num、r0、sig.thres、sig.level、mark等。
-
可映射参数
-
-
r—— 相关系数,适用于geom_square()、geom_circle2()、geom_ellipse2()、geom_pie2()、geom_confbox()、geom_mark()(统计显著性标记的系数,与
1.234∗∗1.234∗∗1.234∗∗
类似)。这些参数之所以都设置为“r”,主要是因为在相关系数可视化中基本都映射为相关系数,统一命名可以减少一些参数记忆,方便使用。
-
p—— 相关系数检验P值,适用于geom_mark()、geom_cross(),结合sig.thres等参数来根据显著性水平做一些辅助标记。 -
r、low、upp—— 适用于geom_confbox(),三个参数均是必须的,low、r、upp分别确定置信区间盒子的下端、中间和上端线条位置。 -
num—— 适用于geom_num(),数值变量。这个函数封装于geom_text(),做了一点点优化,以后可能会删除。
-
-
非映射参数
-
-
r0,外接圆半径缩放系数,适用于geom_square()、geom_circle2()、geom_ellipse2()、geom_pie2()函数。该参数的主要意义是处理图形覆盖问题,当在每个单元格画半径为0.5的方块、圆等图标时,会相互覆盖掉背景网格线,影响视觉效果。该参数默认值是0.48。 -
sig.thres统计显著性临界值,用来过滤非显著的数据行。适用于geom_mark()、geom_cross()。 -
sig.level、mark适用于geom_mark(),前者为统计显著性水平向量(如c(0.001, 0.01, 0.05)),后者为对应的标记符号(c("***", "**", "*"))。统计显著性水平向量不要求一定要按顺序排列,只要求和标记符号一一对应就行。
-
Draw correlation plot quickly
This is a basic example which shows you how to draw correlation plot quickly:
library(ggplot2) library(ggcor) quickcor(mtcars) + geom_colour()
quickcor(mtcars, type = "upper") + geom_circle2()
ggcor(mtcars, type = "lower", show.diag = TRUE) + geom_ellipse2() 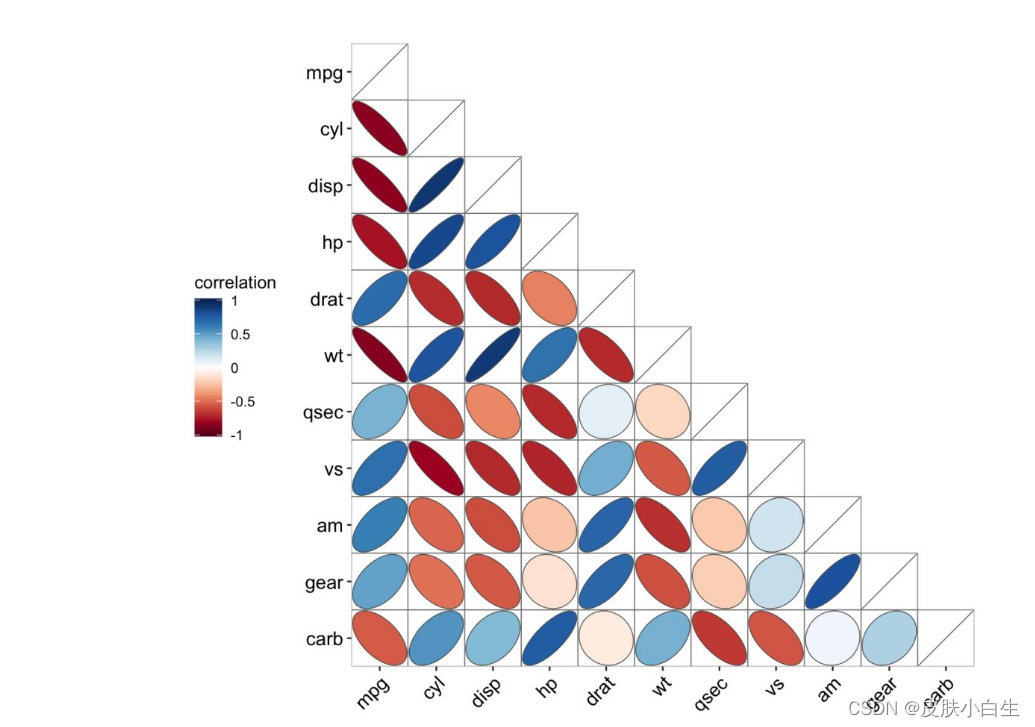
ggcor(mtcars, type = "full", cluster.type = "all") + geom_pie2() ggcor(mtcars, cluster.type = "all") +
geom_colour() +
geom_num(aes(num = r), colour = "grey90", size = 3.5) 
ggcor(mtcars, type = "full", cor.test = TRUE) + geom_confbox() 
quickcor(mtcars, cor.test = TRUE) +
geom_square(data = get_data(type = "lower", show.diag = FALSE)) +
geom_mark(data = get_data(type = "upper", show.diag = FALSE), size = 2.5) +
geom_abline(slope = -1, intercept = 12)Mantel test plot
library(vegan)
library(dplyr)
data("varechem")
data("varespec")
set.seed(20191224)
sam_grp <- sample(paste0("sample", 1:3), 24, replace = TRUE)
mantel01 <- fortify_mantel(varespec, varechem, group = sam_grp,
spec.select = list(spec01 = 1:5,
spec02 = 6:12,
spec03 = 7:18,
spec04 = 20:29,
spec05 = 30:44),
mantel.fun = "mantel.randtest")
quickcor(mantel01, legend.title = "Mantel's r") +
geom_colour() + geom_cross() + facet_grid(rows = vars(.group))
mantel02 <- fortify_mantel(varespec, varechem,
spec.select = list(1:10, 5:14, 7:22, 9:32)) %>%
mutate(r = cut(r, breaks = c(-Inf, 0.25, 0.5, Inf),
labels = c("<0.25", "0.25-0.5", ">=0.5"),
right = FALSE),
p.value = cut(p.value, breaks = c(-Inf, 0.001, 0.01, 0.05, Inf),
labels = c("<0.001", "0.001-0.01", "0.01-0.05", ">=0.05"),
right = FALSE))
quickcor(varechem, type = "upper") + geom_square() +
add_link(mantel02, mapping = aes(colour = p.value, size = r),
diag.label = TRUE) +
scale_size_manual(values = c(0.5, 1.5, 3)) +
add_diag_label() + remove_axis("x")
#> Warning: `add_diag_label()` is deprecated. Use `geom_diag_label()` instead.
network
library(tidygraph)
library(ggraph)
net <- fast_correlate(varespec) %>%
as_tbl_graph(r.thres = 0.5, p.thres = 0.05) %>%
mutate(degree = tidygraph::centrality_degree(mode = "all"))
ggraph(net, "circle") +
geom_edge_fan(aes(edge_width = r, edge_linetype = r < 0),
edge_colour = "grey80", show.legend = FALSE) +
geom_node_point(aes(size = degree, colour = name),
show.legend = FALSE) +
geom_node_text(aes(x = x * 1.08, y = y * 1.08,
angle = -((-node_angle(x, y) + 90) %% 180) + 90,
label = name), size = 4, hjust= "outward",
show.legend = FALSE) +
scale_edge_color_gradientn(colours = c("blue", "red")) +
scale_edge_width_continuous(range = c(0.1, 1)) +
coord_fixed(xlim = c(-1.5, 1.5), ylim = c(-1.5, 1.5)) +
theme_graph()
general heatmap
mat <- matrix(rnorm(120), nrow = 15) cor_tbl(extra.mat = list(mat = mat)) %>% quickcor(mapping = aes(fill = mat)) + geom_colour()
upper and lower with different geom
d <- dist(t(mtcars)) correlate(mtcars, cor.test = TRUE) %>% as_cor_tbl(extra.mat = list(dist = d)) %>% quickcor() + geom_upper_square(aes(upper_fill = r, upper_r0 = r)) + geom_lower_colour(aes(lower_fill = dist)) + geom_diag_label() + remove_all_axis()
【避坑】R报错 lazy-load database is corrupt
即这个错误是暂时的,只要你正确安装R包,重启R session就能解决
![]()
加载包
1)设置工作目录
rm(list=ls())#clear Global Environmentsetwd('D:\\桌面\\mantel test分析')#设置工作路径
2)安装、加载R包
#安装包# install.packages("ggplot2")# install.packages("vegan")# install.packages("dplyr")# install.packages("devtools")# devtools::install_github("houyunhuang/ggcor")#加载包library(vegan)library(dplyr)library(ggcor)library(ggplot2)
![]()
加载数据
#OTU表格df <- read.table("otu.txt",sep="\t",header = T,row.names = 1,check.names = F)df <-data.frame(t(df))#环境因子数据env <- read.table("env.txt",sep="\t",header = T,row.names = 1,check.names = F)head(df)head(env)
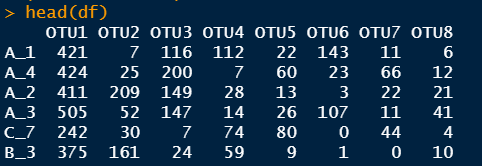
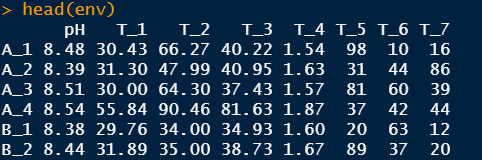
![]()
环境因子的相关性分析及展示
quickcor(env, type = "lower",method = "spearman") +geom_square()+scale_fill_gradient2( high = 'orange', mid = 'white',low = 'navyblue') #颜色设置
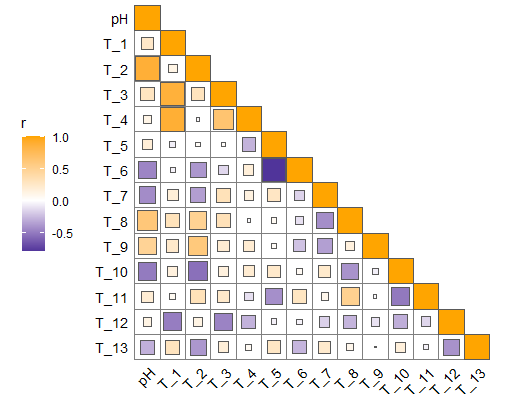
![]()
Mantel test分析
1)计算OTU与环境因子之间的mantel test的r值和p值
df_mantel <- mantel_test(df, env, mantel.fun = 'mantel',spec.dist.method = 'bray',env.dist.method = 'euclidean',spec.select = list(A = 1:4,B = c(6,8,9,10),C = c(5,7,11,12)))#将群落数据按组进行分开
2)定义标签
df_mantel <- df_mantel %>%mutate(df_r = cut(r, breaks = c(-Inf, 0.1, 0.2, 0.4, Inf),labels = c("< 0.1", "0.1 - 0.2", "0.2 - 0.4", ">= 0.4")),#定义Mantel的R值范围标签df_p = cut(p.value, breaks = c(-Inf, 0.01, 0.05, Inf),labels = c("< 0.01", "0.01 - 0.05", ">= 0.05")))#定义Mantel的P值范围标签
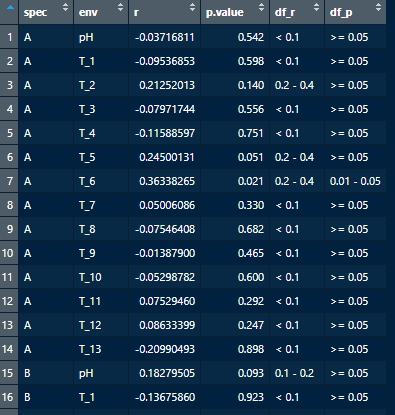
![]()
绘图
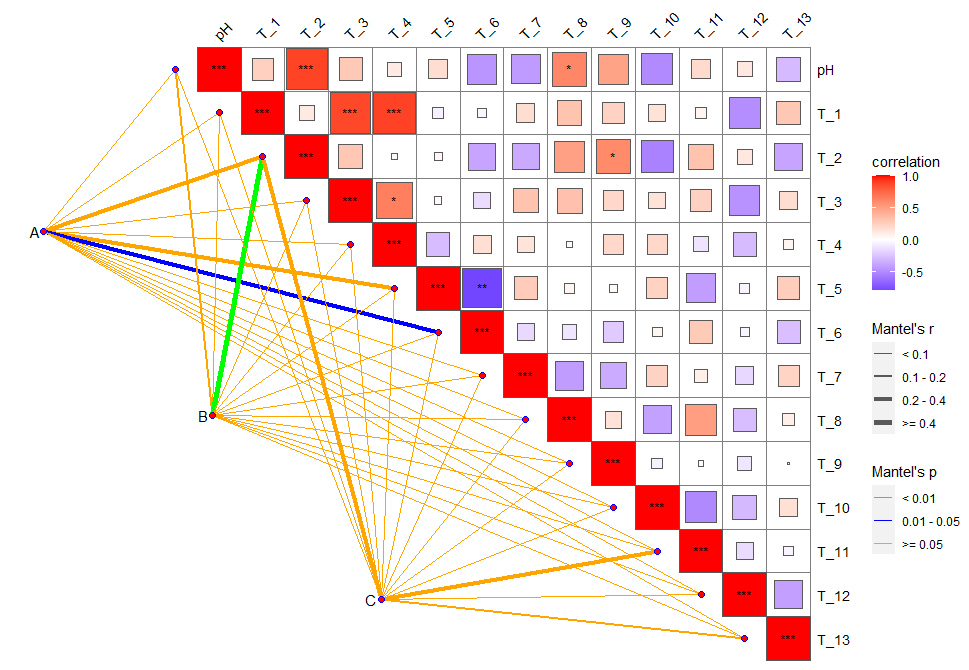
quickcor(env,method = "spearman", type = "upper", cor.test = T, cluster.type = "all") +#环境因子之间的相关性热图geom_square() +#相关性显示形式geom_mark(r = NA,sig.thres = 0.05, size = 3.5, colour = "black")+#显著性标签scale_fill_gradient2( high = 'red', mid = 'white',low = 'blue') + #颜色设置anno_link(df_mantel, aes(color = df_p,size = df_r))+scale_size_manual(values = c(0.5, 1, 1.5, 2))+#连线粗细设置scale_color_manual(values = c("green","blue","orange"))+#线条颜色设置guides(fill = guide_colorbar(title = "correlation", order = 1),#图例相关设置size = guide_legend(title = "Mantel's r",order = 2),color = guide_legend(title = "Mantel's p", order = 3),linetype = "none")
参考:
1)https://zhuanlan.zhihu.com/p/507384776
2)https://zhuanlan.zhihu.com/p/110501058
-
ggcor(mtcars, type = "full", cor.test = TRUE, cluster.type = "all") + -
geom_colour() + geom_cross()
-
ggcor(mtcars, type = "full", cor.test = TRUE) + -
geom_square() + geom_cross()
其它图层函数的使用都比较符合直觉,需要设置的地方也很少,而geom_mark()会涉及一些其它问题,这里拉出来说说。
很多时候,我们并不关心不具备统计显著性的相关系数,也不需要在图中显示,这时需要设置sig.thres,即要过滤的显著性临界值。
-
ggcor(mtcars, type = "full", cor.test = TRUE, cluster.type = "all") + -
geom_raster() + -
geom_mark(sig.thres = 0.05, size = 3, colour = "grey90")
若是只要统计显著性标记的"*"号,不要系数怎么整?在geom_mark()中也很简单,直接设置r就好。
-
ggcor(mtcars, type = "full", cor.test = TRUE, cluster.type = "all") + -
geom_raster() + -
geom_mark(r = NA, sig.thres = 0.05, size = 5, colour = "grey90")
那要改变星号标记规则,只要小于0.05的标记一颗星,其它什么都不标记呢?注意:因为星号在文本中显示在偏上的位置,若不设置vjust参数,看上去纵向会不居中。
-
ggcor(mtcars, type = "full", cor.test = TRUE, cluster.type = "all") + -
geom_raster() + -
geom_mark(r = NA, sig.level = 0.05, mark = "*", vjust = 0.65, size = 6, colour = "grey90")
非对称相关系数矩阵
非对称相关系数矩阵和非对称矩阵是有细微的区别的,前者表示行列代表不同的变量集合,相互之间的顺序可以打乱。所以,有时候要分析两个表中每个变量之间的相关性,此时得到的结果就是非对称的相关系数矩阵。
-
library(vegan) # 使用vegan包所带的数据集 -
data(varechem) -
data(varespec) -
df03 <- fortify_cor(x = varechem, y = varespec[ , 1:30], cluster.type = "col") -
ggcor(df03) + geom_colour()
-
df04 <- fortify_cor(x = varespec[ , 1:30], y = varechem, cor.test = TRUE) -
ggcor(df04) + geom_square() + geom_cross(size = 0.2)# 数据集不好,没几个显著的
上下三角不一样怎么画?
ggcor提供了很多辅助函数来对cor_tbl数据进行过滤,函数命名规则上都以get_*开头。
-
get_lower_data()—— 获取相关系数矩阵下三角所在行,仅支持对称的相关系数矩阵。 -
get_upper_data()—— 获取相关系数矩阵上三角所在行,仅支持对称的相关系数矩阵。 -
get_diag_data()—— 获取相关系数矩阵对角线所在行,仅支持对称的相关系数矩阵。 -
get_diag_tri()—— 删除相关系数矩阵对角线所在行,仅支持对称的相关系数矩阵。 -
get_data()—— 是以上四个函数的重新包装,主要在画图时使用,稍后通过例子详细说明。
-
df05 <- fortify_cor(x = varechem, cor.test = TRUE, cluster.type = "all") -
ggcor(df05) + geom_circle2()
-
df05_lower <- get_lower_data(df05, show.diag = FALSE) -
ggcor(df05_lower) + geom_circle2()
-
ggcor(df05) + -
geom_pie2(data = get_data(type = "upper", show.diag = FALSE)) + -
geom_ellipse2(data = get_data(type = "lower", show.diag = TRUE))
-
ggcor(df05) + -
geom_segment(aes(x = x - 0.5, y = y + 0.5, xend = x + 0.5, yend = y - 0.5), -
data = get_data(type = "diag"), size = 0.5, colour = "grey60") + -
geom_colour(data = get_data(type = "upper", show.diag = FALSE)) + -
geom_mark(data = get_data(type = "upper", show.diag = FALSE), size = 3) + -
geom_circle2(data = get_data(r >= 0.5, type = "lower", show.diag = FALSE), -
r = 0.8, fill = "#66C2A5") + -
geom_num(aes(num = r), data = get_data(type = "lower", -
show.diag = FALSE), size = 3)
列名放在对角线?
-
ggcor(mtcars, cor.test = TRUE, cluster.type = "all") + -
geom_confbox(data = get_data(type = "upper", show.diag = FALSE)) + -
geom_num(aes(num = r), data = get_data(type = "lower", show.diag = FALSE), size = 3.5) + -
add_diaglab(size = 4.56) + remove_axis()
想对颜色分组?
很多情况下,连续性颜色棒并不是很好分区每个单元格对应的数值区间,这时根据相关系数大小对颜色进行分组可能更适合。说来非常巧合,一直头疼这个问题难以解决,就在前不久,ggraph包的作者Thomas Lin向ggplot2包提交了一组新的颜色映射函数(scale_fill/colour_steps*()),这个问题迎刃而解,非常幸运。
ggcor()函数有参数fill.binned,默认为FALSE,设置为TRUE就会根据相关系数大小对颜色分组。若要控制分组的数量和区间,可以通过legend.breaks来设置。
ggcor(mtcars, fill.bin = TRUE) + geom_square() # 默认分组-
ggcor(mtcars, fill.bin = TRUE, legend.breaks = seq(-1, 1, length.out = 11)) + -
geom_square() #指定分组,0.2为一个区间
-
col <- col <- ggcor:::.default_colors -
col[6] <- "#F2F2F2" -
ggcor(mtcars, cluster.type = "all", fill.colours = col, fill.bin = T, -
legend.breaks = c(-1, -0.8, -0.5, 0.5, 0.8, 1)) + -
geom_colour()
玩点花活
这部分内容要在线下载表情,很多时候会因为网络问题下载失败。不给图了。
-
library(ggimage) -
emoji <- c("1f004", "1f0cf", "1f170", "1f171", "1f17e", -
"1f17f", "1f18e", "1f191", "1f192", "1f193", -
"1f194", "1f195", "1f196", "1f197") -
ggcor(df05) + -
geom_pokemon(aes(image=ifelse(r > 0.5, 'pikachu', 'tauros')), -
data = get_data(type = "lower", show.diag = FALSE)) + -
geom_emoji(aes(image = ifelse(p <= 0.05, '1f600', '1f622')), -
data = get_data(type = "upper", show.diag = FALSE)) + -
geom_emoji(aes(image = emoji), data = get_data(type = "diag"))
-
ggcor(df05) + -
geom_pokemon(aes(image=ifelse(r > 0.5, 'pikachu', 'tauros')), -
data = get_data(type = "lower", show.diag = FALSE)) + -
geom_colour(data = get_data(type = "upper", show.diag = FALSE)) + -
geom_shade(data = get_data(type = "upper", show.diag = FALSE), -
sign = -1, size = 0.1) + -
geom_emoji(aes(image = emoji), data = get_data(type = "diag"))
mantel 检验组合图
mantel 检验(Mantel test 是对两个矩阵相关关系的检验)的组合图已经十分流行了,用各种工具做的都有。大概5月份的时候,我基于corrplot模拟重现了那幅图,直到现在每周都有人询问我相关实现的问题,我基本都是回答说等新方案,因为那个实现很复杂,没有基本的R知识,很难替换成自己的数据。8月底,我在ggcorrr(ggcor前身)开发了gghpcc,几乎可以满足可视化的要求,但是在优化ggcor的过程中,我才恍然大悟,mantel检验本质上也是相关性分析,何不统一整合进ggcor呢,就这样,gghpcc被干掉了。
进行案例展示之前,我先解释一个关键性问题,这个问题在目前框架下没有很好的办法去自动化解决方案,那就是坐标轴范围,需要手动设置。前文有提及,这里再重复一次,ggcor()初始化的默认坐标范围是和correlation matrix的行列数相关的,若行数为n,列数为m,x轴范围是c(0.5, m + 0.5),y轴的范围是c(0.5, n + 0.5)。
数据预处理函数
ggcor提供了mantel检验的封装函数fortify_mantel(),支持vegan包中的mantel()、mantel.partial()和ade4包中的mantel.randtest()、mantel.rtest()函数,差别上说mantel.partial()是偏mantel检验(有控制变量),其它三个是mantel检验,当不使用并行计算时,mantel.randtest()速度最快(底层是C语言),mantel.rtest()最慢,纯粹R代码实现。
-
spec是物种数据,支持列表(list,非data.frame)或者数据框(data.frame)。 -
-
若是列表,列表中每个元素构成一个群落;
-
若是数据框(最常见的情况),数据框中的每一列是一个物种(OTU),每行是一个样本,可以通过
spec.select参数来指定哪些列构成一个群落。例如若spec中1-4列是spec01群落,5-12是spec02群落,spec.select = list(spec01 = 1:4, spec02 = 5:12)(当然,你也可以(最好不)不指定群落名称,这是名字自动命名)。还有一种情况(设置spec.group参数的情况)后面单独解释。
-
-
env是环境数据,支持列表(list,非data.frame)或者数据框(data.frame),env中的每个元素对应一个环境变量(当然,若是列表,也可以支持多个环境变量组合成一个环境因素的情况)。还有一种情况(设置env.group参数的情况)后面单独解释。 -
env.ctrl是环境控制变量,支持列表(list,非data.frame)或者数据框(data.frame)。需要注意,当env.ctrl非列表时,每次计算的控制环境是相同的,若需要分别设置不同的控制环境,需要通过列表手动设置。还有一种情况(设置env.ctrl.group参数的情况)后面单独解释。 -
mantel.funmantel函数,“mantel”、“mantel.randtest”、“mantel.rtest”和“mantel.partial”。 -
spec.dist.fun物种距离函数,vegdist或者dist。 -
env.dist.fun环境距离函数,vegdist或者dist。注意,当前的情况下,控制环境使用环境距离函数。 -
spec.dist.method物种距离计算方法,参数默认是“bray”。 -
env.dist.method环境距离计算方法,参数默认是“euclidean”。 -
...其他传递给mantel.fun的参数。
group相关的参数是为了处理需要根据样本进行分组的情况,比如我A、B、C三个不同的样本分组,物种、环境和控制环境(均必须为数据框)同样如此,可以通过向量索引(和样本量等长)来指定分组。这时,每个样本组的物种只和对应样本组的环境列表的每个元素进行mantel测试。注意:以上情况均需要设置is.pair = TRUE。
-
## function(spec, -
## env, -
## env.ctrl = NULL, -
## mantel.fun = "mantel", -
## is.pair = FALSE, -
## spec.select = NULL, # a list of index vector -
## spec.group = NULL, -
## env.group = NULL, -
## env.ctrl.group = NULL, -
## spec.dist.fun = "vegdist", -
## env.dist.fun = "vegdist", -
## spec.dist.method = "bray", -
## env.dist.method = "euclidean", -
## ...)
案例一:按列设置物种群落
-
library(vegan) # 使用vegan包所带的数据集 -
data(varechem) -
data(varespec) -
fortify_mantel(varespec, varechem, spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20))
-
## # A tibble: 56 x 4 -
## spec env r p -
## <chr> <chr> <dbl> <dbl> -
## 1 spec01 N 0.100 0.129 -
## 2 spec02 N 0.254 0.011 -
## 3 spec03 N 0.0596 0.238 -
## 4 spec04 N 0.182 0.038 -
## 5 spec01 P 0.165 0.028 -
## 6 spec02 P 0.112 0.129 -
## 7 spec03 P -0.0240 0.573 -
## 8 spec04 P 0.201 0.02 -
## 9 spec01 K 0.0481 0.295 -
## 10 spec02 K 0.307 0.008 -
## # … with 46 more rows
案例二:按样本分组
-
group <- rep(LETTERS[1:3], 8) -
fortify_mantel(varespec[ , 38:43], varechem, -
spec.group = group, -
env.group = group, -
is.pair = TRUE, -
mantel.fun = "mantel.randtest")
-
## # A tibble: 42 x 4 -
## spec env r p -
## <chr> <chr> <dbl> <dbl> -
## 1 A N -0.334 0.884 -
## 2 B N 0.212 0.167 -
## 3 C N 0.241 0.223 -
## 4 A P -0.295 0.888 -
## 5 B P 0.250 0.099 -
## 6 C P 0.312 0.14 -
## 7 A K 0.0909 0.31 -
## 8 B K 0.310 0.101 -
## 9 C K 0.0720 0.33 -
## 10 A Ca -0.365 0.942 -
## # … with 32 more rows
案例三:偏mantel测试
-
fortify_mantel(varespec, varechem[ , 1:10], env.ctrl = varechem[ , 11:14], -
spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20), -
mantel.fun = "mantel.randtest")
-
## # A tibble: 40 x 4 -
## spec env r p -
## <chr> <chr> <dbl> <dbl> -
## 1 spec01 N 0.100 0.134 -
## 2 spec02 N 0.254 0.017 -
## 3 spec03 N 0.0596 0.243 -
## 4 spec04 N 0.182 0.039 -
## 5 spec01 P 0.165 0.031 -
## 6 spec02 P 0.112 0.122 -
## 7 spec03 P -0.0240 0.582 -
## 8 spec04 P 0.201 0.009 -
## 9 spec01 K 0.0481 0.271 -
## 10 spec02 K 0.307 0.007 -
## # … with 30 more rows
mantel检验可视化
mantel检验既然是相关性分析的一种特殊情况,那么相关性分析的可视化也同样适用于mantel检验。当然,由于fortify_mantel()返回数据类和fortify_cor()返回的数据类并不一样,需要经过一道转换程序。
-
mantel <- fortify_mantel(varespec, varechem, spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20)) -
df06 <- as_cor_tbl(mantel) -
ggcor(df06) + geom_pie2() + geom_cross()
mantel检验组合图
mantel组合图是与相关性分析高度整合的,依赖于相关性分析函数,换句话说mantel组合图只是在相关性分析图的基础上额外叠加了一个图层。核心函数是add_link(),是个泛型函数(后面另说)。参数暂时不解释了,看例子。实在抱歉,add_mantel()又被我干掉了。
-
corr <- fortify_cor(varechem, type = "upper", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
mantel <- fortify_mantel(varespec, varechem, -
spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20), -
mantel.fun = "mantel.randtest") -
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(mantel, diag.label = TRUE) + -
add_diaglab(angle = 45) + -
geom_square() + remove_axis("y")
-
corr <- fortify_cor(varechem, type = "upper", show.diag = FALSE, -
cor.test = TRUE, cluster.type = "all") -
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(mantel, diag.label = TRUE) + -
add_diaglab(angle = 45) + -
geom_pie2() + remove_axis("y")
-
corr <- fortify_cor(varechem, type = "lower", show.diag = FALSE, -
cor.test = TRUE, cluster.type = "all") -
ggcor(corr, xlim = c(0.5, 20)) + -
add_link(mantel, diag.label = TRUE) + -
add_diaglab(angle = 45) + -
geom_ellipse2() + remove_axis("y")
-
## 仅是测试效果,没有实际意义 -
corr <- fortify_cor(varechem, varechem[ , 1:7], type = "full", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
mantel <- fortify_mantel(varespec, varechem, -
spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20), -
mantel.fun = "mantel.randtest", nrepet = 2000) -
extra.params <- extra_params(group.label = text_params(size = 6), -
link.params = link_params(group.point.hjust = 2)) -
ggcor(corr, axis.y.position = "left", legend.position = "left", xlim = c(0.5, 14.5)) + -
add_link(mantel, extra.params = extra.params) + -
geom_circle2()
-
group <- rep(LETTERS[1:3], 8) -
corr <- fortify_cor(varechem, type = "upper", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
mantel <- fortify_mantel(varespec[ , 38:43], varechem, -
spec.group = group, -
env.group = group, -
is.pair = TRUE, -
mantel.fun = "mantel.randtest") -
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(mantel, diag.label = TRUE) + -
add_diaglab(angle = 45) + -
geom_colour() + geom_shade(sign = -1) + -
remove_axis("y")
![]()
-
corr <- fortify_cor(varechem, type = "upper", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
mantel <- fortify_mantel(varespec, varechem, -
spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20), -
mantel.fun = "mantel.randtest") -
mantel <- dplyr::filter(mantel, p <= 0.05) -
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(mantel, diag.label = TRUE, legend.drop = TRUE) + -
add_diaglab(angle = 45) + -
geom_square() + remove_axis("y")
## 个人觉得很丑
只要简单的相关性?
-
corr <- fortify_cor(varechem, type = "upper", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
corr01 <- fortify_cor(varechem, varespec[ , 38:39], type = "upper", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
mantel <- fortify_mantel(varespec, varechem, -
spec.select = list(spec01 = 22:25, -
spec02 = 1:4, -
spec03 = 38:43, -
spec04 = 15:20), -
mantel.fun = "mantel.randtest") -
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(x = corr01, diag.label = TRUE, -
link.line.colours = c("#E31A1C", "#33A02C")) + -
add_diaglab(angle = 45) + -
geom_square() + remove_axis("y")
![]()
-
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(x = corr01, mapping = aes(size = abs(r)), diag.label = TRUE) + -
add_diaglab(angle = 45) + -
geom_square() + -
scale_size_continuous(limits = c(0, 1), range = c(0.25, 3)) + -
guides(size = guide_legend(title = "abs r", override.aes = list(colour = "grey35"), -
order = 1)) + -
remove_axis("y")
更灵活的方案
若要自己更灵活的处理,或者是要用这个组合图展示其它类型的数据,add_link()支持自定义的数据框做参数。第一个参数df需要一个数据框,包含x和y列,x列类似于mantel检验中的物种群落(或者是样本组),y类似于mantel检验中的环境变量。在内部,会自动用df中的y和相关系数矩阵的行名进行匹配确定坐标位置。
-
corr <- fortify_cor(varechem, type = "upper", show.diag = TRUE, -
cor.test = TRUE, cluster.type = "all") -
df <- data.frame(x = rep(LETTERS[1:3], 14), -
y = rep(cor_tbl_yname(corr), 3), -
r = runif(42, -1, 1), -
p = runif(42, 0, 0.5), stringsAsFactors = FALSE) -
ggcor(corr, xlim = c(-5, 14.5)) + -
add_link(df, diag.label = TRUE, colour = "red") + -
add_diaglab(angle = 45) + -
geom_square() + remove_axis("y")
相关性网络图
这块内容不会整合在ggcor包里面,但是利用ggcor里面的函数很容易导出相关性分析数据供其它函数使用。
-
df07 <- fortify_cor(varespec, type = "upper", show.diag = FALSE, -
cor.test = TRUE, keep.name = TRUE) -
df07
-
## # A tibble: 946 x 6 -
## x y r p low upp -
## <fct> <fct> <dbl> <dbl> <dbl> <dbl> -
## 1 Empenigr Callvulg -0.262 0.217 -0.602 0.159 -
## 2 Rhodtome Callvulg -0.0608 0.778 -0.453 0.351 -
## 3 Rhodtome Empenigr 0.529 0.00781 0.160 0.769 -
## 4 Vaccmyrt Callvulg -0.0925 0.667 -0.478 0.323 -
## 5 Vaccmyrt Empenigr 0.205 0.337 -0.216 0.562 -
## # … with 941 more rows -
## Extra attributes: -
## xname: Callvulg Empenigr Rhodtome Vaccmyrt Vaccviti Pinusylv ... -
## yname: Cladphyl Claddefo Cladcerv Icmaeric Peltapht Stersp N ... -
## show.diag: FALSE
-
## make graph -
library(ggraph) -
library(tidygraph) -
graph <- as_tbl_graph(df07) -
ggraph(graph, layout = 'linear', circular = TRUE) + -
geom_edge_arc(aes(colour = r, alpha = p)) + -
scale_edge_alpha_continuous(range = c(1, 0.1)) + -
coord_fixed() #颜色是相关性,线条浓淡是统计检验P值
智能推荐
oracle 12c 集群安装后的检查_12c查看crs状态-程序员宅基地
文章浏览阅读1.6k次。安装配置gi、安装数据库软件、dbca建库见下:http://blog.csdn.net/kadwf123/article/details/784299611、检查集群节点及状态:[root@rac2 ~]# olsnodes -srac1 Activerac2 Activerac3 Activerac4 Active[root@rac2 ~]_12c查看crs状态
解决jupyter notebook无法找到虚拟环境的问题_jupyter没有pytorch环境-程序员宅基地
文章浏览阅读1.3w次,点赞45次,收藏99次。我个人用的是anaconda3的一个python集成环境,自带jupyter notebook,但在我打开jupyter notebook界面后,却找不到对应的虚拟环境,原来是jupyter notebook只是通用于下载anaconda时自带的环境,其他环境要想使用必须手动下载一些库:1.首先进入到自己创建的虚拟环境(pytorch是虚拟环境的名字)activate pytorch2.在该环境下下载这个库conda install ipykernelconda install nb__jupyter没有pytorch环境
国内安装scoop的保姆教程_scoop-cn-程序员宅基地
文章浏览阅读5.2k次,点赞19次,收藏28次。选择scoop纯属意外,也是无奈,因为电脑用户被锁了管理员权限,所有exe安装程序都无法安装,只可以用绿色软件,最后被我发现scoop,省去了到处下载XXX绿色版的烦恼,当然scoop里需要管理员权限的软件也跟我无缘了(譬如everything)。推荐添加dorado这个bucket镜像,里面很多中文软件,但是部分国外的软件下载地址在github,可能无法下载。以上两个是官方bucket的国内镜像,所有软件建议优先从这里下载。上面可以看到很多bucket以及软件数。如果官网登陆不了可以试一下以下方式。_scoop-cn
Element ui colorpicker在Vue中的使用_vue el-color-picker-程序员宅基地
文章浏览阅读4.5k次,点赞2次,收藏3次。首先要有一个color-picker组件 <el-color-picker v-model="headcolor"></el-color-picker>在data里面data() { return {headcolor: ’ #278add ’ //这里可以选择一个默认的颜色} }然后在你想要改变颜色的地方用v-bind绑定就好了,例如:这里的:sty..._vue el-color-picker
迅为iTOP-4412精英版之烧写内核移植后的镜像_exynos 4412 刷机-程序员宅基地
文章浏览阅读640次。基于芯片日益增长的问题,所以内核开发者们引入了新的方法,就是在内核中只保留函数,而数据则不包含,由用户(应用程序员)自己把数据按照规定的格式编写,并放在约定的地方,为了不占用过多的内存,还要求数据以根精简的方式编写。boot启动时,传参给内核,告诉内核设备树文件和kernel的位置,内核启动时根据地址去找到设备树文件,再利用专用的编译器去反编译dtb文件,将dtb还原成数据结构,以供驱动的函数去调用。firmware是三星的一个固件的设备信息,因为找不到固件,所以内核启动不成功。_exynos 4412 刷机
Linux系统配置jdk_linux配置jdk-程序员宅基地
文章浏览阅读2w次,点赞24次,收藏42次。Linux系统配置jdkLinux学习教程,Linux入门教程(超详细)_linux配置jdk
随便推点
matlab(4):特殊符号的输入_matlab微米怎么输入-程序员宅基地
文章浏览阅读3.3k次,点赞5次,收藏19次。xlabel('\delta');ylabel('AUC');具体符号的对照表参照下图:_matlab微米怎么输入
C语言程序设计-文件(打开与关闭、顺序、二进制读写)-程序员宅基地
文章浏览阅读119次。顺序读写指的是按照文件中数据的顺序进行读取或写入。对于文本文件,可以使用fgets、fputs、fscanf、fprintf等函数进行顺序读写。在C语言中,对文件的操作通常涉及文件的打开、读写以及关闭。文件的打开使用fopen函数,而关闭则使用fclose函数。在C语言中,可以使用fread和fwrite函数进行二进制读写。 Biaoge 于2024-03-09 23:51发布 阅读量:7 ️文章类型:【 C语言程序设计 】在C语言中,用于打开文件的函数是____,用于关闭文件的函数是____。
Touchdesigner自学笔记之三_touchdesigner怎么让一个模型跟着鼠标移动-程序员宅基地
文章浏览阅读3.4k次,点赞2次,收藏13次。跟随鼠标移动的粒子以grid(SOP)为partical(SOP)的资源模板,调整后连接【Geo组合+point spirit(MAT)】,在连接【feedback组合】适当调整。影响粒子动态的节点【metaball(SOP)+force(SOP)】添加mouse in(CHOP)鼠标位置到metaball的坐标,实现鼠标影响。..._touchdesigner怎么让一个模型跟着鼠标移动
【附源码】基于java的校园停车场管理系统的设计与实现61m0e9计算机毕设SSM_基于java技术的停车场管理系统实现与设计-程序员宅基地
文章浏览阅读178次。项目运行环境配置:Jdk1.8 + Tomcat7.0 + Mysql + HBuilderX(Webstorm也行)+ Eclispe(IntelliJ IDEA,Eclispe,MyEclispe,Sts都支持)。项目技术:Springboot + mybatis + Maven +mysql5.7或8.0+html+css+js等等组成,B/S模式 + Maven管理等等。环境需要1.运行环境:最好是java jdk 1.8,我们在这个平台上运行的。其他版本理论上也可以。_基于java技术的停车场管理系统实现与设计
Android系统播放器MediaPlayer源码分析_android多媒体播放源码分析 时序图-程序员宅基地
文章浏览阅读3.5k次。前言对于MediaPlayer播放器的源码分析内容相对来说比较多,会从Java-&amp;gt;Jni-&amp;gt;C/C++慢慢分析,后面会慢慢更新。另外,博客只作为自己学习记录的一种方式,对于其他的不过多的评论。MediaPlayerDemopublic class MainActivity extends AppCompatActivity implements SurfaceHolder.Cal..._android多媒体播放源码分析 时序图
java 数据结构与算法 ——快速排序法-程序员宅基地
文章浏览阅读2.4k次,点赞41次,收藏13次。java 数据结构与算法 ——快速排序法_快速排序法

