使用wgd进行全基因组复制分析-程序员宅基地
因为全基因组复制(Whole genome duplications, WGD)是生物进化的重要因素之一, 所以WGD分析也是基因组分析经常用到的一种分析方法。举个例子,我们之所以能在多条染色体之间发现一些古老基因连锁现象,是因为被子植物在过去2亿年时间里就出现了多次的全基因组复制和基因丢失事件(见下图,Tang et al., 2008)
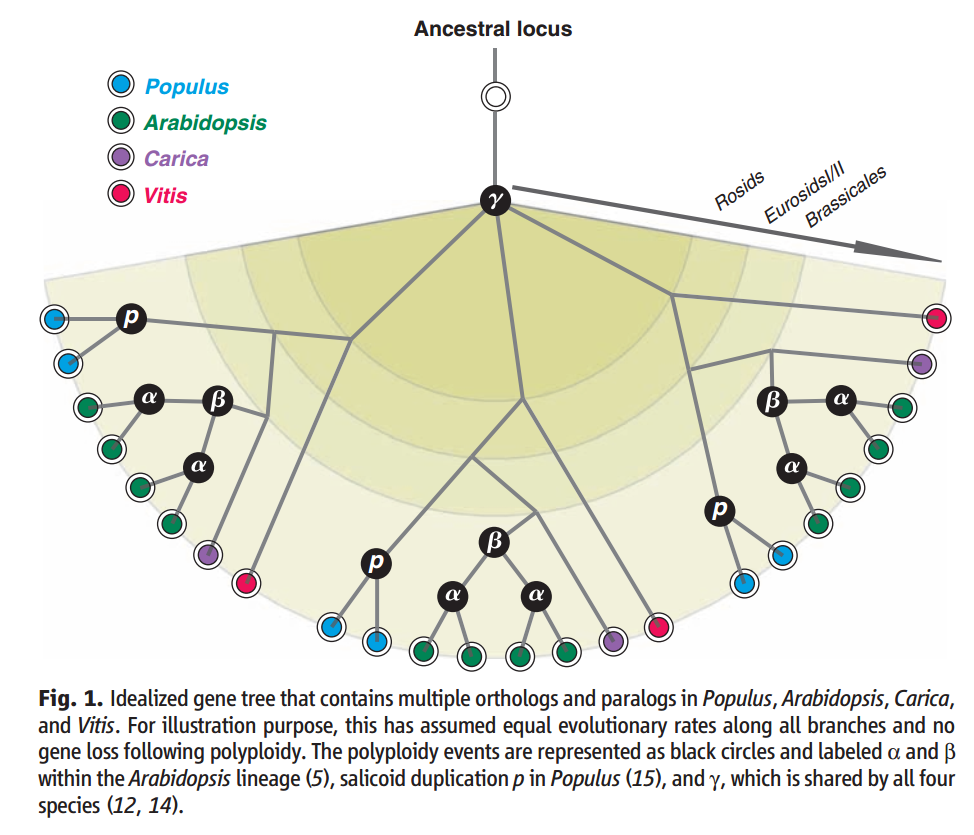
古老WGD检测有两种方法,一种是共线性分析,另一种则是根据Ks分布图。其中Ks定义为平均每个同义位点上的同义置换数,与其对应的还有一个Ka,指的是平均每个非同义位点上的非同义置换数。
如果没有WGD或是大片段重复,那么基因组中的旁系同源基因的同义置换符合指数分布(exponential distribution), 反之,Ks分布图中就会出现一个由于WGD导致的正态分布峰(normal distributed peak). 而古老WGD的年龄则可通过分析这些峰中的同源置换数目来预测(Tiley et al., 2018)。
以发表在Science上的Papaver somniferum L. 基因组文章中的图Fig 1C为例,文章分别分析了P. somniferum 和其他几个物种的Ks分布。从Ks分布图可以看到P. somniferum经历了一次比较近的WGD事件(Guo et al., 2018)。
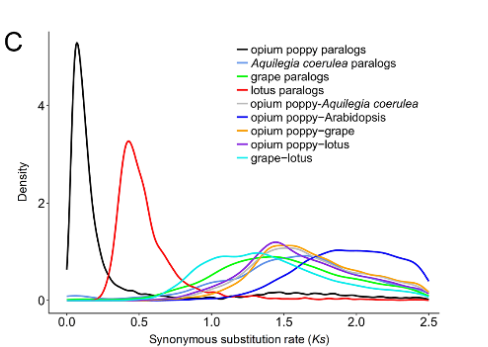
文章中关于WGD的分析流程参考附录8.1 Whole genome duplication in opium poppy genome, 我总结了关键的几点
- 使用megablast进行自比对,寻找基因组中共线性的区块
- 使用BLASTP基于RBH( reciprocal best hits )进行蛋白之间的相互比对
- 使用KaKs_Calculator基于YN模型计算RBH基因对中的Ks(synonymous substitution rate)
- 为了区分Ks中peak是WGD事件还是小规模重复,作者使用MCScanX进行共线性分析,发现93.9%的RBH基因都是基因组内共线性
目前WGD的分析流程也有人发了文章,我通过关键字"wgd pipeline"搜索找到了如下几个:
- GenoDup: https://github.com/MaoYafei/GenoDup-Pipeline
- WGDdetector: https://github.com/yongzhiyang2012/WGDdetector
- wgd: https://github.com/arzwa/wgd
这一篇介绍的是wgd的用法。
软件安装
wgd目前无法用bioconda直接安装,所以安装起来稍显麻烦,这是因为它的依赖软件有点多。wgd依赖于以下软件
- BLAST
- MCL
- MUSCLE/MAFFT/PRANK
- PAML
- PhyML/FastTree
- i-ADHoRe
但是好消息是它依赖的软件大部分都可以用bioconda进行安装
conda create -n wgd python=3.5 blast mcl muscle mafft prank paml fasttree cmake libpng mpi=1.0=mpich
conda activate wgd
这里选择了mpi=1.0=mpich, 原因是i-adhore依赖于mpich. 如果安装了openmpi就会出现error while loading shared libraries: libmpi_cxx.so.40: cannot open shared object file: No such file or directory
之后的安装就简单多了
git clone https://github.com/arzwa/wgd.git
cd wgd
pip install .
# 或者一行命令
pip install git+https://github.com/arzwa/wgd.git
对于i-ADHoRe,需要先在http://bioinformatics.psb.ugent.be/webtools/i-adhore/licensing/同意许可,才能下载i-ADHoRe-3.0
由于我的miniconda3安装在~/opt/下,所以安装路径为~/opt/miniconda3/envs/wgd/
tar -zxvf i-adhore-3.0.01.tar.gz
cd i-adhore-3.0.01
mkdir -p build && cd build
cmake .. -DCMAKE_INSTALL_PREFIX=~/opt/miniconda3/envs/wgd/
make -j 4
make insatall
软件介绍
WGD一共有9个子模块
- kde: 对Ks分布进行KDE拟合
- ksd : Ks分布构建
- mcl:All-vs-ALl的BLASP比对 + MCL分类分析.
- mix: Ks分布的混合建模.
- pre: 对CDS文件进行预处理
- syn: 调用I-ADHoRe 3.0利用GFF文件进行共线性分析
- viz: 绘制柱状图和密度图
- wf1: 全基因组旁系同源基因组(paranome)的Ks标准分析流程,调用mcl, ksd和syn
- wf2: one-vs-one 同源基因(ortholog)的Ks标准分析流程,调用wcl和ksd
流程示意图如下:
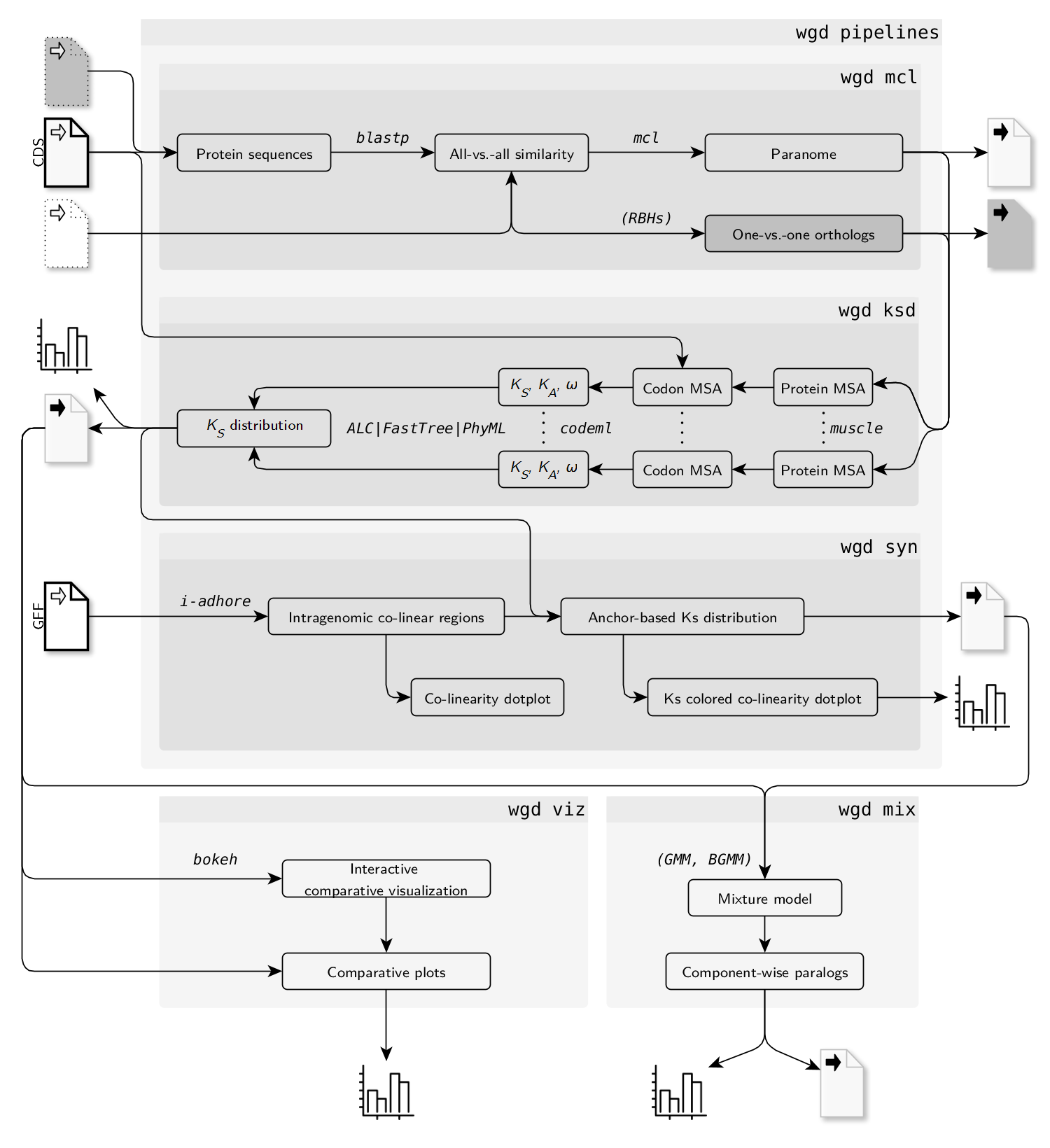
使用方法
以甘蔗的基因组 Saccharum spontaneum L 为例,基因组为8倍体,共32条染色体(2n = 4x8 = 32)
下载CDS和GFF注释文件 tutorial
mkdir -p wgd_tutorial && cd wgd_tutorial
wget http://www.life.illinois.edu/ming/downloads/Spontaneum_genome/Sspon.v20190103.cds.fasta.gz
wget http://www.life.illinois.edu/ming/downloads/Spontaneum_genome/Sspon.v20190103.gff3.gz
gunzip *.gz
先用conda activate wgd启动我们的分析环境,然后就开始分析了
第一步: 使用wgd mcl鉴定基因组内的同源基因
wgd mcl -n 20 --cds --mcl -s Sspon.v20190103.cds.fasta -o Sspon_cds.out
# -n: 线程
# --cds: 输入为cds
# --mcl: mcl聚类
这一步运行过程中,wgd会先检查cds序列是否有效,也就是是否以ATG(起始密码子)开头,且以TAA/TAG/TGA(终止密码子)结尾,然后将cds转成氨基酸序列,之后用Blastp进行相互比对,然后根据blastp结果用mcl聚类的方式寻找旁系同源基因。
输出结果在Sspon_cds.out,有两个结果输出
- blast.tsv: BLASTP的outfmt6输出结果
- blast.tsv.mcl: MCL聚类结果,每一行可以认为是一个基因家族(gene family)
第二步: 使用wgd ksd构建Ks分布
wgd ksd --n_threads 80 Sspon_cds.out/Sspon.v20190103.cds.fasta.blast.tsv.mcl Sspon.v20190103.cds.fasta
这一步也是先过滤cds中的无效数据,然后用mafft(默认)/muscle/prank对每个基因家族进行多重序列联配,用codeml计算dN/dS, 用alc/fasttree(默认)/phyml建树
输出结果在wgd_ksd目录下
- ks.tsv: 每个基因家族中基因对的各项统计,其中包括Ka和Ks
- ks.svg: Ks分布,见下图
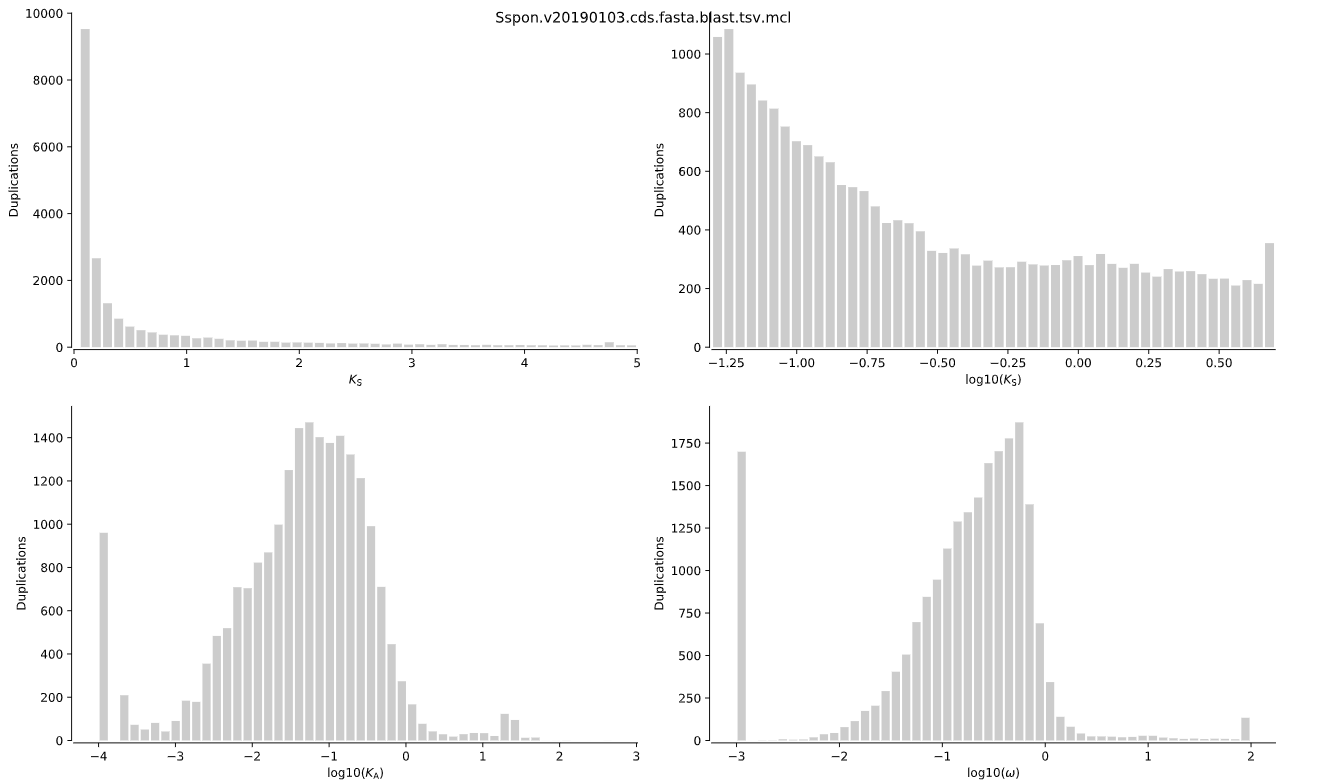
第三步: 如果基因组质量不错,那么可以使用wgd syn进行共线性分析。它能帮我们找到基因组内的共线性区块,以及相应的锚定点
wgd syn --feature gene --gene_attribute ID \
-ks wgd_ksd/Sspon.v20190103.cds.fasta.ks.tsv \
Sspon.v20190103.gff3 Sspon_cds.out/Sspon.v20190103.cds.fasta.blast.tsv.mcl
#--feature: 从第三列选择特征
#--gene_attribute: 从第九列提取编号
输出图片以.svg结尾,如下所示,图中颜色红蓝代表Ks得分。
<img src="http://xuzhougeng.top/upload/2019/9/wgd_syn-169aae6bd15d4ed192d245da884c88a0.png" alt="wgd syn" style="zoom:50%;" />
Ks的下游分析通常还包括对Ks分布的统计建模,这可以使用wgd kde进行核密度拟合(Kernel density estimate, KDE)或用wgd mix建立高斯混合模型(Gaussian mixture models)
# KDE
wgd kde wgd_ksd/Sspon.v20190103.cds.fasta.ks.tsv
# Gaussian
wgd mix wgd_ksd/Sspon.v20190103.cds.fasta.ks.tsv
wgd kde输出kde.svg, 而wdg mix则生成一个 wgd_mix文件夹。
混合模型常用于基于Ks分布研究WGD。基于一些基本的分子进化假设,我们预期Ks分布中由WGD导致的peak应该符合高斯分布,能够用log-normal分布近似。但是考虑到混合模型容易过拟合,特别是Ks分布,因此作者不建议使用混合模型作为多次WGD假设的正式统计测试。
参考文献
Tang, H., Bowers, J.E., Wang, X., Ming, R., Alam, M., and Paterson, A.H. (2008). Synteny and Collinearity in Plant Genomes. Science 320, 486–488.
Tiley, G.P., Barker, M.S., and Burleigh, J.G. (2018). Assessing the Performance of Ks Plots for Detecting Ancient Whole Genome Duplications. Genome Biol Evol 10, 2882–2898.
Guo, L., Winzer, T., Yang, X., Li, Y., Ning, Z., He, Z., Teodor, R., Lu, Y., Bowser, T.A., Graham, I.A., et al. (2018). The opium poppy genome and morphinan production. Science 362, 343–347.
Zwaenepoel, A., and Van de Peer, Y. (2019). wgd—simple command line tools for the analysis of ancient whole-genome duplications. Bioinformatics 35, 2153–2155.
智能推荐
分卷压缩与分卷解压_分段压缩和解压-程序员宅基地
分卷压缩与分卷解压是一种用于文件压缩和解压的技术。该技术可以将大文件分成多个较小的卷,并且可以分别对每个卷进行压缩和解压缩操作。这种技术在处理大型文件时非常有用,可以提高文件传输的效率和方便性。
【前端资源分享】推荐收藏的前端学习资源_前端分享内容-程序员宅基地
文章浏览阅读545次,点赞7次,收藏12次。今天分享一些个人收藏的前端学习资源,按一下几个维度简单划分了下,有 3D、框架、构建工具等等。由于这些地址都是我个人收藏的,所以带有一些强烈的主观意识,还有很多优秀的网址没有收录进来,会不断更新的,欢迎大家点赞,收藏。_前端分享内容
android studioapp成功图,AndroidStudio多渠道打包当你完成了一个app项目,后面发现不同客户需要定制不同ui,或者功能,这个时候怎么办? 拿ui来说,第一种方法,不同客户替...-程序员宅基地
文章浏览阅读223次。最近不断有朋友向我咨询AndroidStudio多渠道的打包方法,今天整理一下之前积累的打包套路,写一篇文章,手把手的教给大家。 说到多渠道,这里不得不提一下友盟统计,友盟统计是大家日常开发中常用的渠道统计工具,而我们的打包方法就是基于友盟统计实施的。按照友盟官方文档说明,渠道信息通常需要在AndroidManifest.xml中配置如下值:上面的value值Channel_ID就是..._android studio多渠道res
python 使用HTMLReport生成测试报告-程序员宅基地
文章浏览阅读8.3k次。一、安装: 在线安装:使用pip命令安装HTMLReport 命令:pip install HTMLReport 安装好的位置在:Python安装路径下的Lib/site-packages下 离线安装:直接将下载好的HTMLR..._htmlreport
前端实现打印预览功能以及page-break-inside属性解决打印换行问题(打印预览表格或文字被分割开)-程序员宅基地
文章浏览阅读1.4w次,点赞4次,收藏26次。遇到的问题:打印预览的时候表格被分割了,就是一共两页而其中一行显示在不同的两个页面。如下图:_page-break-inside
稀疏矩阵在matalb中_spconvert函数-程序员宅基地
文章浏览阅读298次。1.存储2.转化为稀疏矩阵 sparse函数和full函数3.直接建立稀疏矩阵 spconvert函数4.带状稀疏矩阵spdiags函数例题:求解三对角线方程组的解_spconvert函数
随便推点
哈工大硕士生用 Python 实现了 11 种经典数据降维算法,源代码库已开放_std_broken = broken.std(axis=0)-程序员宅基地
文章浏览阅读3k次,点赞4次,收藏86次。导语:适合机器学习初学者和刚入坑数据挖掘的小伙伴雷锋网 AI 开发者按:网上关于各种降维算法的资料参差不齐,同时大部分不提供源代码。这里有个 GitHub 项目整理了使用 Python 实现了 11 种经典的数据抽取(数据降维)算法,包括:PCA、LDA、MDS、LLE、TSNE 等,并附有相关资料、展示效果;非常适合机器学习初学者和刚刚入坑数据挖掘的小伙伴。为什么要进行数据降维?..._std_broken = broken.std(axis=0)
建议收藏 | 数据化、信息化、数字化、智能化到底都是指什么?彼此有什么联系?_数据化智能化的象征代表-程序员宅基地
文章浏览阅读7.5k次,点赞2次,收藏31次。随着新技术、新产业、新业态、新模式的不断出现,各行各业的企业都在寻找新的突破口进行转型升级,“数据化、信息化、数字化、智能化”愈来愈频繁地出现在大众视野中,关于它们概念和解说也是层出不穷、百花齐放,到底它们之间有什么区别呢? Runwise整理了一些关于数据化、信息化、数字化、智能化的相关定义,结合组织定义与行业发展趋势,对四者之间的联系与区别进行解析,便于大家更好理解之间的关系。01 关于数据化、信息化、数字化、智能化的概念数据化1.数据化的定义数据代表着对某一件事物的描述,通过记录、分析、重组数_数据化智能化的象征代表
pdf转word用python轻松搞定_使用Python将PDF转化为word-程序员宅基地
文章浏览阅读1.6k次。60行Python代码,实现多线程PDF转Word分解任务把PDF转为Word,分几步?两步,第一步读取PDF文件,第二步写入Word文件。是的,就是这么简单,借助Python第三方包,可以轻松实现上面两个过程,我们要用到pdfminer3k和python-docx这两个包读取PDFfrom pdfminer.pdfinterp import PDFResourceManagerfrom pdfm..._python写pdf与word互转代码
fmea手册_新版FMEA打分怎么破?(详细收录手册标准对照表...-程序员宅基地
文章浏览阅读5.6k次。新版FMEA终于正式发布了!关于新版FMEA正式版与草稿版的差异,后续黄老师将会每周撰写新文来给大家做解读及分享,敬请持续关注公众号(首页菜单的历史文章中有FMEA合集,大家可随时点击阅读旧文)。为了方便部分已经懂了七步法的学员可以直接进行新版FMEA的使用,本篇特别将新版FMEA手册中的评分表整理出来,方便参考使用。本篇文章建议收藏,后续可持续做为工具书随时参考使用。关于打分说起FMEA,打分是..._fmea打分标准对照表
Fantastical 2 for Mac(日历管理软件) v2.5免激活版-程序员宅基地
文章浏览阅读3.3k次。今天小编为您带来Fantastical 2 Mac一款易于使用的日历管理软件,Fantastical 2 Mac版采用了全新的设计风格,和Yosemite系统十分贴合,并且提供了「光」和“黑暗”两种配色模式,可以切换左栏的颜色。右侧的布局和系统原生日历十分相像,而在左侧则显示了该月日期及行程安排,并且还整合了系统原生的 「提醒事项」 。Fantastical 2Mac破解版使用说明下载完成...
微信小程序|自定义弹窗组件_微信小程序的自定义弹窗-程序员宅基地
文章浏览阅读1.3w次,点赞63次,收藏66次。深入探讨如何创建自定义弹出组件,穿插案例研究和实际应用,在实际开发中更好地利用自定义弹出组件来提升用户体验。_微信小程序的自定义弹窗